Proyecto de secuencia genómica de un Saccharomyces cerevisiae aislado natural de Colombia
El bioetanol es un biocombustible ecológico, limpio y renovable que se fusiona como una opción para descartar el impacto ambiental de los combustibles fósiles. Los microorganismos predominantes responsables de la producción eficiente de etanol son cepas seleccionadas de la levadura Saccharomyces cerevisiae.
En este trabajo se caracteriza el genoma de una cepa aislada natural de S. cerevisiae que se seleccionó de un grupo de levaduras debido a su potencial para producir etanol de primera y segunda generación, comparable con otras cepas aisladas y comerciales naturales (como Red Fermentis ® ). Esta cepa tiene la capacidad innata de consumir, a baja velocidad, la xilosa sin modificación. Por estos motivos, generamos un ensamblaje y una anotación de novo para Saccharomyces cerevisiaeisolate 202-3, y brindamos ideas para ingeniería metabólica para la producción de bioetanol a partir de biomasa.
Con el borrador de la secuencia del genoma, quisimos averiguar si nuestra cepa tiene la capacidad de consumir xilosa debido a la presencia de algunos genes descubiertos en algunas cepas de S. cerevisiae , principalmente como levadura de vino y contribuir con información de la variabilidad genética de la levadura S. cerevisiae.
Variedad de levadura y medios de crecimiento
El S. cerevisiae 202-3 aislado natural se seleccionó de un grupo de levaduras bien caracterizadas por su producción de bioetanol a partir de hidrolizado de bagazo de caña de azúcar. La corroboración del uso de xilosa como única fuente de carbono se realizó en medio YNBX de agar (xilosa 5%, agar 2%, base de nitrógeno de levadura 0,67%) en condiciones estándar. Las células de levadura se cultivaron rutinariamente en medio YPD (extracto de levadura al 1%, peptona al 2% y glucosa al 2%) en condiciones estándar.
Análisis de la secuencia del genoma
El ADN genómico se aisló de las células de la cepa 202-3 con columnas YeaStar (Zymo Research) de acuerdo con las recomendaciones del fabricante.
La secuenciación del genoma de escopeta se realizó utilizando el extremo pareado de Illumina Hiseq 2000. La evaluación de la calidad se realizó con FastQC , y la limpieza basada en la puntuación de calidad se realizó con el software Prinseq.
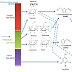
Se realizó un montaje de novo utilizando el software SOAPdenovo versión 2.04. Kmer tamaño de 61 se utilizó para realizar el proceso.
Las predicciones genéticas se realizaron mediante el enfoque del modelo oculto de Markov (HMM), utilizando AUGUSTUS, con la cepa de referencia S288c de S. cerevisiae . Se realizó una anotación automática utilizando el algoritmo BLASTp comparando las proteínas predichas extraídas con las proteínas de S. cerevisiae de la base de datos del genoma de Saccharomyces (SGD).
Finalmente, los identificadores de proteínas anotadas se utilizaron como entradas para la Base de datos de anotación, visualización y descubrimiento integrado (DAVID) para obtener categorías de clasificación general.
Resultados
El proceso de secuenciación generó los archivos de lectura de extremo pareado R1 y R2, que consisten en un total de 21 '730.524 lecturas. El conjunto resultante tiene 120 andamios con una longitud total de 12'855.005 pb, un N50 de 330 kb y un contenido de GC del 38%.
La anotación del genoma realizada por AUGUSTUS reveló 5.366 marcos de lectura abierta putativos (ORF), que eran proteínas predichas potenciales. Una anotación automática utilizando el algoritmo BLASTp reveló 5.230 proteínas potenciales con similitud significativa.
El análisis de DAVID reveló la presencia de genes comunes informados en S. cerevisiae S 288c en el metabolismo de la xilosa. Para confirmar la presencia del gen que confiere la capacidad de crecimiento en la xilosa, se realizó un BLASTx con los genes reportados del metabolismo de la xilosa en algunas cepas de S. cerevisiae , con especial atención en el gen XDH1, que confiere Capacidad de crecimiento en xilosa a algunas cepas. Estos datos revelan que la cepa 202-3 tiene un fondo genético potencial beneficioso para la producción efectiva de bioetanol a partir de fuentes de carbono C6 y C5, sin la presencia del gen XDH1. Los análisis genómicos de 202-3 son, por lo tanto, esenciales para un mayor refinamiento de las vías metabólicas de la xilosa.
FUENTE: IEEE
En este trabajo se caracteriza el genoma de una cepa aislada natural de S. cerevisiae que se seleccionó de un grupo de levaduras debido a su potencial para producir etanol de primera y segunda generación, comparable con otras cepas aisladas y comerciales naturales (como Red Fermentis ® ). Esta cepa tiene la capacidad innata de consumir, a baja velocidad, la xilosa sin modificación. Por estos motivos, generamos un ensamblaje y una anotación de novo para Saccharomyces cerevisiaeisolate 202-3, y brindamos ideas para ingeniería metabólica para la producción de bioetanol a partir de biomasa.
Con el borrador de la secuencia del genoma, quisimos averiguar si nuestra cepa tiene la capacidad de consumir xilosa debido a la presencia de algunos genes descubiertos en algunas cepas de S. cerevisiae , principalmente como levadura de vino y contribuir con información de la variabilidad genética de la levadura S. cerevisiae.
Variedad de levadura y medios de crecimiento
El S. cerevisiae 202-3 aislado natural se seleccionó de un grupo de levaduras bien caracterizadas por su producción de bioetanol a partir de hidrolizado de bagazo de caña de azúcar. La corroboración del uso de xilosa como única fuente de carbono se realizó en medio YNBX de agar (xilosa 5%, agar 2%, base de nitrógeno de levadura 0,67%) en condiciones estándar. Las células de levadura se cultivaron rutinariamente en medio YPD (extracto de levadura al 1%, peptona al 2% y glucosa al 2%) en condiciones estándar.
Análisis de la secuencia del genoma
El ADN genómico se aisló de las células de la cepa 202-3 con columnas YeaStar (Zymo Research) de acuerdo con las recomendaciones del fabricante.
La secuenciación del genoma de escopeta se realizó utilizando el extremo pareado de Illumina Hiseq 2000. La evaluación de la calidad se realizó con FastQC , y la limpieza basada en la puntuación de calidad se realizó con el software Prinseq.
Se realizó un montaje de novo utilizando el software SOAPdenovo versión 2.04. Kmer tamaño de 61 se utilizó para realizar el proceso.
Las predicciones genéticas se realizaron mediante el enfoque del modelo oculto de Markov (HMM), utilizando AUGUSTUS, con la cepa de referencia S288c de S. cerevisiae . Se realizó una anotación automática utilizando el algoritmo BLASTp comparando las proteínas predichas extraídas con las proteínas de S. cerevisiae de la base de datos del genoma de Saccharomyces (SGD).
Finalmente, los identificadores de proteínas anotadas se utilizaron como entradas para la Base de datos de anotación, visualización y descubrimiento integrado (DAVID) para obtener categorías de clasificación general.
Resultados
El proceso de secuenciación generó los archivos de lectura de extremo pareado R1 y R2, que consisten en un total de 21 '730.524 lecturas. El conjunto resultante tiene 120 andamios con una longitud total de 12'855.005 pb, un N50 de 330 kb y un contenido de GC del 38%.
La anotación del genoma realizada por AUGUSTUS reveló 5.366 marcos de lectura abierta putativos (ORF), que eran proteínas predichas potenciales. Una anotación automática utilizando el algoritmo BLASTp reveló 5.230 proteínas potenciales con similitud significativa.
El análisis de DAVID reveló la presencia de genes comunes informados en S. cerevisiae S 288c en el metabolismo de la xilosa. Para confirmar la presencia del gen que confiere la capacidad de crecimiento en la xilosa, se realizó un BLASTx con los genes reportados del metabolismo de la xilosa en algunas cepas de S. cerevisiae , con especial atención en el gen XDH1, que confiere Capacidad de crecimiento en xilosa a algunas cepas. Estos datos revelan que la cepa 202-3 tiene un fondo genético potencial beneficioso para la producción efectiva de bioetanol a partir de fuentes de carbono C6 y C5, sin la presencia del gen XDH1. Los análisis genómicos de 202-3 son, por lo tanto, esenciales para un mayor refinamiento de las vías metabólicas de la xilosa.
FUENTE: IEEE











No hay comentarios.: